RCSB PDBのJmol表示がver.11.6になった影響
昨日突然起こった大事件。標題のように,RCSB PDBで採用している分子モデル表示形式のうち,Jmolがver.11.6に変更された。
機能がかなり増え,モデル上でマウス右クリックして表示されるメニューも以下のように変更された。まずは表示構造データをその場で保存できるようになったことは大きい。
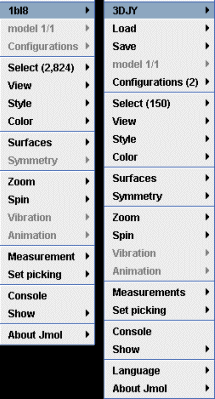
Jmolの表示変更メニュー:〔左〕Jmol 11.0.2(本サイト),〔右〕Jmol 11.6
昨日はなぜかRCSB PDBでは“File Not Found”エラーで分子モデルが表示されたのだが今日は正常になった。
- データ例(昨日の新規公開データの中から):3DJY
この表示を見てわかるように,データの下にはJmol scriptを入力できるフォームが新登場した。早速,私のJmolコンテンツで使っているscriptを入れて(ソースからコピペ)ボタンを押すとちゃんと表示変更できるではないですか。
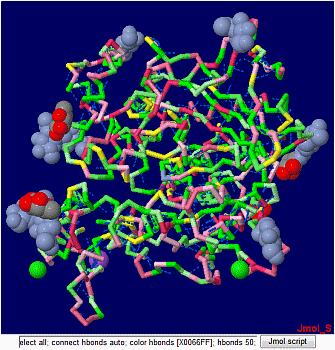
3DJYを有機概念図I/O値順着色,糖鎖は糖別着色し,水素結合表示
これを楽しんでもらうために,タンパク質について有機概念図簡易計算機の雰囲気を実感できるscriptを以下に示そう。
- select all; wireframe off; backbone off; spacefill; select asn or ser; colour atoms [255,60,120]; select asp or gln; colour atoms [255,120,160]; select glu or thr; colour atoms [255,160,200]; select arg or his; colour atoms [255,200,220]; select gly; colour atoms [255,255,50]; select lys or tyr or trp; colour atoms [170,255,170]; select cys or met; colour atoms [130,255,130]; select pro or phe; colour atoms [100,255,100]; select ala or val or ile or leu; colour atoms [0,255,0];
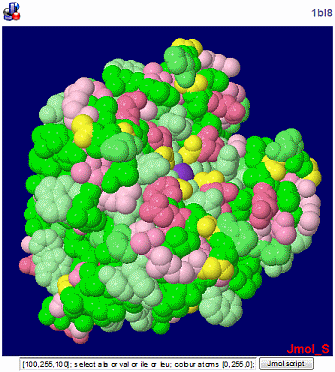
カリウムチャネルの例1BL8の表示変更例(リガンドはCPK色)
※有機概念図簡易計算機参照
Jmol 11.6でいろいろ遊べそうだが,その時間が少しないのが残念。是非いろいろ試みてこの記事にコメントやTBをお願いしたい。