無性生殖集団の親子鑑定
- 倍々で増える無性生殖生物集団を考える
- すべての個体は不死であって、増殖を続けると仮定する
- DNA配列はポアッソン乱数で変異が入る
- Infinite modelを仮定し、変異は必ず新規なものとする
- グラフオブジェクトに納めることで
- グラフ上の距離(パスの長さ)が親等に
- エッジに変異の数の重みを与えて、グラフ上の重み付き距離(重み付きパス長)を取れば、それが、DNA配列の異なる塩基数になる
- このような単純な集団において、親等と配列の違いとの関係を考慮することは、生殖集団におけるDNA配列の差による、親等の鑑別の(もっとも)基礎的な枠組みに相当する
- Rでのグラフ理論(igraphパッケージ)についての基礎はこちらを
library(igraph) # 無性生殖生物の親子鑑定 # 倍々で増える # 変異は増えるだけ infinite site model Ngen<-6 poisl<-5 # グラフのエッジリストである2列の行列 edges<-NULL # グラフのエッジの重みを格納するベクトル edge.weight<-NULL cnt<-1 #v<-c(0) for(i in 1:Ngen){ # 変異数はポアッソン乱数 rs<-rpois(cnt*2,poisl) #v<-c(v,rep(v,2)+rs) edges<-rbind(edges,matrix(c(rep(1:cnt,2),(cnt+1):(cnt*3)),ncol=2)-1) edge.weight<-c(edge.weight,rs) cnt<-cnt+length(rs) } # グラフオブジェクト化 g<-graph.edgelist(edges) # グラフをプロット plot(g,layout=layout.circle) # ノード間の距離(親等に相当) sh<-shortest.paths(g) # ノード間の配列の違う箇所数は重み付き距離 sh.weight<-shortest.paths(g,weights=edge.weight) #dna.dist<-abs(outer(v,v,FUN="+")) # 親等の最小・最大 path.len.range<-range(sh) # ノード間の親等別ペア数 hist(c(sh)) # ノード間の配列の違いの度数分布 hist(c(sh.weight)) # 親等ごとに配列の違いを度数分布 par(ask=TRUE) dist.per.kinship<-list() for(i in 1:(path.len.range[2]-path.len.range[1]+1)){ dist.per.kinship[[i]]<-sh.weight[which(sh==path.len.range[1]+i-1)] hist(dist.per.kinship[[i]]) } # 親等別に配列の違いをボックスプロット boxplot(dist.per.kinship) par(ask=FALSE)
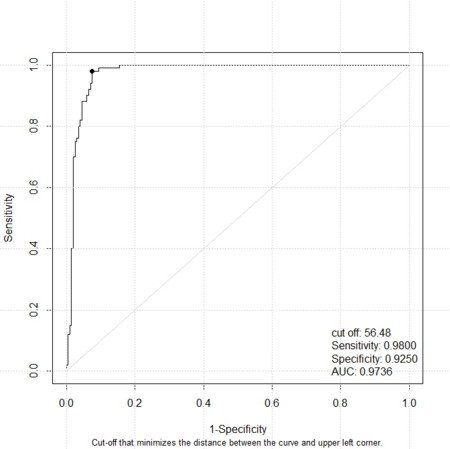
DiagnosisMedパッケージを使ってROCカーブを描いてみる
- こちらを参考
library(DiagnosisMed) Ncase<-100 Ncont<-100 mean.case<-40 sd.case<-3 mean.cont<-35 sd.cont<-2 Case<-rnorm(Ncase,mean.case,sd.case) Cont<-rnorm(Ncont,mean.cont,sd.cont) boxplot(cbind(Case,Cont)) ra<-range(c(Case,Cont)) breaks<-seq(from=floor(ra[1]),to=ceiling(ra[2]),by=0.5) h.cont<-hist(Cont,breaks=breaks,density=20) h.case<-hist(Case,breaks=breaks,col=2,density=30) ylim<-c(0,max(h.cont$counts,h.case$counts)) plot(h.cont,ylim=ylim,density=20) par(new=TRUE) plot(h.case,ylim=ylim,col=2,density=30) V<-c(Case,Cont) Pheno<-c(rep(1,Ncase),rep(0,Ncont)) ROC(Pheno,V)
カットオフ
- こちらの企画
- 親子鑑定
- 親子かそうじゃないか(親子ではなくて、『ただの関係』)
- 尤度の比をとる
- ルール・インする尤度比の基準がある
- ルール・アウトする尤度比の基準がある
- 尤度比検定
- パラメタがあって、仮説がパラメタで表現されている
- 仮説ごとに尤度が計算できる
- 尤度の比の対数を取ったものと、カイ自乗統計量の間には、パラメタ数の差〜自由度 な関係がある
- カットオフを決める
- 感度、特異度、PPV、NPVが決まる
- この3者の関係を上手に説明してみたい
- 仮説はいくつある?
- 仮説を分類する(家系図上の距離で分類)?
- 標本に観察された情報によって、仮説空間が変わる?
- 感度・特異度がある?
- PPV・NPVがある?
- 感度・特異度・PPV・NPVのすべてがある?すべてはない?
- 尤度比だけが、「基準」?
- 尤度分布がパラメタの作る「ユークリッドな空間」にあるわけではなくて、「グラフ的な空間」にあったり、非ユークリッド空間にあったりするってことはない?
- DNAを用いて、「多型」以外に情報が取れない?